Humilde e diria primária tradução de:
W. Ford Doolittle and Olga Zhaxybayeva; Evolution: Reducible Complexity — The Case for Bacterial Flagella; Current Biology; Volume 17, Issue 13, 3 July 2007, Pages R510-R512; doi:10.1016/j.cub.2007.05.003
Defensores do Design Inteligente (D.I.) afirmam que algumas estruturas biológicas são "irredutivelmente complexas", formadas por partes que não podem ser úteis por si mesmas, e requerem para sua disposição um designer inteligente. O flagelo bacteriano é uma de tais estruturas, o equivalente do século XXI ao olho vertebrado - a origem do que Darwin mesmo admitia como sendo um teste para sua teoria. Argumentos sobre como um flagelo poderia ter sido composto, passo a passo, a partir de proteínas antecedentes que tinha em cada fase de algum tipo de utilização parcial ou alternativas foram percebidas como grandes argumentos no julgamento de 2005, em Dover, Pennsylvania, sobre o ensino de D.I. como ciência em escolas públicas.
Esse julgamento foi contra os antievolutionistas, o juiz Jones colocando de que a teoria dominante do D.I. "não pode dissociar-se de seu criacionismo e, portanto, possui antecedentes religiosos". Esta decisão não tem, é claro, como acabar com o movimento criacionista, por isso os esforços dos evolucionistas em elaborar e testar cenários evolutivos para flagelos continuam a ser moeda política, bem como de interesse científico.
Distribuição das proteínas flagelares. (T. Ryan Gregory; Genome sequences reduce the complexity of bacterial flagella. - www.genomicron.evolverzone.com - April 16th, 2007
Em outubro passado, Pallen e Matzke [1] resumiram em uma revisão muito do conhecimento relevante. Flagelos bacterianos são, de fato, diversos em composição (e bastante distintos dos análogos archaea), mas no referente a oito proteínas bacterianas axiais esses autores inferem que "o complexo haste-gancho- flagelo claramente evoluiu por várias rodadas de duplicação gênica e posterior diversificação , a partir de apenas duas proteínas (uma proto-flagelina e uma proteína proto-haste/gancho) ". Há também relações de homologia em muitas proteínas não-flagelares. Como Matzke coloca (comunicação pessoal): "de 42 assim chamadas proteínas flagelares padrão em Escherichia coli / Salmonella, apenas 20 são universalmente necessárias / detectáveis em todas as flagelos, apenas 15 não têm homólogos conhecidos, e apenas duas são ambas necessárias e universalmente não têm homólogos." Então, o palco parece montado para uma contagem mais detalhada dos eventos, funções intermediárias e as pressões seletivas que dão origem a essas estruturas incríveis.
Um grande passo nessa direção já foi reivindicado por Liu e Ochman [2]. O artigo rapidamente atraiu comentários (favoráveis) no ScienceNOW [3] e desencadeou uma tempestade de comentários (a favor e contra) na blogosfera evolutiva. No ScienceNOW, Michael Lynch é citado como dizendo “Complexidade constrói-se da simplicidade, e isso [o artigo de Liu e Ochman] é um argumento bem documentado de como isto pode ter acontecido.” Talvez sim, mas há algumas ressalvas nós, evolucionistas, devemos considerar antes de saudar Liu e Ochman [2] como os nossos próximos campeões na guerra contra a irracionalidade. É importante que examinemos os seus argumentos com especial cuidado, porque eles tendem a estar sob contenção no próximo julgamento.
Liu e Ochman [2] apresentam duas conclusões sobre as histórias evolucionárias dos 24 genes ‘nucleares’ [Nota do tradutor 1] do flagelo em bactérias que eles consideram ancestrais para todas as bactérias flageladas. Primeiro, eles afirmam que todos os 24 (e não apenas oito) são homólogos entre si, derivando de um único ancestral através de duplicações sucessivas e diversificações, uma sequência de eventos que reconstruem com análises filogenéticas. Segundo, eles argumentam que a transferência lateral de genes (LGT, lateral gene transfer) tem desempenhado apenas um papel menor na evolução desses 24 genes, que com apenas duas exceções "cada um dos genes tem seguido uma história comum em bactérias, uma vez que eles tenham se originado" (presumivelmente em um tempo inicial, antes da divergência das principais linhagens bacterianas).
Nota do tradutor 1: Aqui, traduzi "core" (miolo, caroço, núcleo, âmago) como núcleo e adjetivei, embora, mesmo com ganho em sentido e qualidade do texto, sei que gero a confusão com núcleo celular, mas acredito que serei perdoado por meus leitores biólogos e afins pois me parece óbvio que não poderia, em se tratando de bactérias, tratar-se de núcleo com o sentido de estrutura celular/organela.
A evidência apresentada para a primeira reivindicação é a pontuação BLAST aparentemente significativa entre muitos genes flagelares individuais, coletivamente unindo todos eles. Essa hipótese de "ancestral único para todas as proteínas nucleares flagelares" é, no entanto, fortemente criticada no blog da Panda Thumb (http://www.pandasthumb.org/) por Matzke, que sugere que configuração falha de omissões de BLAST enganaram Liu e Ochman [2], e que, para além das homologias entre proteínas axiais já observadas são mal interpretadas. Igualmente problemática, pensamos, é a sua conclusão de que "as proteínas que formam o flagelo, as proteínas de haste, gancho e filamentos, originados em uma ordem que reflete o processo de montagem flagelar "de dentro para fora". O senso comum pode sugerir um cenário como esse, mas apenas árvores enraizadas, que Liu e Ochman [2] não fornecem, podem prová-lo.
Existe evidência mais sólida para a congruência de histórias evolutivas individuais de genes nucleares, em que o segundo pedido (pequeno LGT) se baseia? Liu e Ochman [2] chegam a sua conclusão, comparando bem suportados ramos (aqueles com suporte de bootstrap > 75%) para cada uma das 24 árvores de genes individuais contra bem suportadas ramificações na árvore de 14 genes fundamentais universalmente presentes, concatenados (encadeados) e tratados como um único gene. Inspecção em detalhe de seus dados mostra que o número de posições alinháveis para conjuntos de dados de genes individuais (gentilmente fornecidos por R. Liu) é frequentemente muito baixa (tão baixo como sete aminoácidos) e as árvores filogenéticas resultantes, em geral, a falta de suporte de bootstrap alto para a maioria dos ramos indivíduais. Cinco das 24 árvores de genes núcleares tem apenas um ramo bem suportado, e os conjuntos de genes com muitos ramos apoiados muitas vezes têm vários homólogos por organismo, fazendo com que muitos ramos suportados sejam irrelevante para a comparação com a árvore de referência concatenada. Se apenas muitos poucos ramos estão de acordo com a árvore de referência (e muitas vezes estes são ramos diferentes em cada conjunto de genes), então não há realmente nenhuma evidência de que eles compartilham uma "história comum" (sem LGT) em bactérias.
Críticos da LGT frequentemente assumem estas árvores com pouca resolução, porque elas não mostram conflito estatisticamente significativo, devem estar de acordo e apoiam a descendência. Mas na maioria das vezes não é simplesmente sinal insuficiente. Fazendo a descendência a hipótese nula é se assumir o que era para ser provado - e, neste caso para dar credibilidade ao falso credibilidade a uma história evolutiva comum. Além disso, sete dos 24 genes realmente discordam significativamente com a filogenia concatenada para um ou dois ramos. Assim, Liu e Ochman [2] muito provavelmente esqueceram alguns eventos documentáveis da LGT, bem como confundindo sistematicamente "ausência de evidências" para a LGT como "evidência de ausência" da LGT.
Este mesmo viés pode ser responsável pela interpretação errônea da comparação da árvore filogenética concatenada de 14 genes para uma árvore de 'espécies' de referência (reconstruído a partir principalmente das proteínas ribossômicas [4]), que é mostrada como a sua Figura 2 (ver abaixo). Embora Liu e Ochman [2] afirmem que apenas quatro histórias de táxons exibem diferentes, as duas árvores filogenéticas realmente discordam em 13 ramos (contando apenas aquelas com mais de 75% de apoio bootstrap) embora concordando com apenas 10. Além da taxa quatro realçada como afetados por LGT nesta figura, existem conflitos que envolvem as posições de Desulfotalea psychrophila, Idiomarina loihiensis, Gluconobacter oxydans, Caulobacter crescentus e o grupo Mesorhizobium que compreende, Sinorhizobium e Agrobacterium, bem como os conflitos de ramos internos .
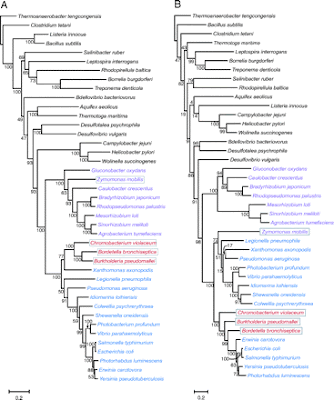 |
| A figura (2) do artigo de Liu and Ochman. [2] |
Antes de Darwin, o argumento do design - eloquentemente encapsulado no aforismo de William Paley deque o relógio no brejo - parecia convincente. Desde Darwin, a seleção natural operando oportunamente na variação forneceu uma alternativa naturalista, tornando desnecessário invocar design consciente para explicar a adaptação. Na verdade mesmo D.I.-istas aceitam explicações darwinistas para adaptações que considerem menos que irredutivelmente complexas. Então nós, evolucionistas, não precisamos assumir o desafio impossível de fixar para baixo todos os detalhes da evolução flagelar. Nós precisamos apenas mostrar que esse desenvolvimento, envolvendo processos e componentes não muito diferentes daquelas que já conhecemos e podemos concordar, é viável. O cenário específico sugerido por Liu e Ochman [2] - passo-a-passo da elaboração de uma estrutura de finalidade única complexa usando apenas a duplicação e divergência dentro de uma linhagem única do genoma - é particularmente onerosa, e coincidentemente especialmente vulnerável a erros de interpretação por aqueles que precisam ver a evolução como intencional. Alternativas envolvendo LGT, remendando peças com origens distintas e múltiplas em outras funções originais poderiam tirar proveito de combinatória, princípios de mistura e acerto, e facilitarem a evolução da complexidade. Então, se Liu e Ochman [2] virem a estar errados sobre a extensão da LGT, este não deve ser motivo de D.I.-istas para alegrarem-se.
Referências
1. M.J. Pallen and N.J. Matzke, From The Origin of Species to the origin of bacterial flagella, Nat. Rev. Microbiol. 4 (2006), pp. 784–790. PDF / Abstract
2. R. Liu and H. Ochman, Stepwise formation of the bacterial flagellar system, Proc. Natl. Acad. Sci. USA 104 (2007), pp. 7116–7121. Abstract
3. Cutraro, J. (2007). A complex tail, simply told. ScienceNOW Daily News.
4. F.D. Ciccarelli, T. Doerks, C. von Mering, C.J. Creevey, B. Snel and P. Bork, Toward automatic reconstruction of a highly resolved tree of life, Science 311 (2006), pp. 1283–1287.
Leitura recomendada
- Songye Chen, et al.; Structural diversity of bacterial flagellar motors; The EMBO Journal 30, 2972 - 2981 (14 June 2011) | doi:10.1038/emboj.2011.186
- Renyi Liu and Howard Ochman; Origins of Flagellar Gene Operons and Secondary Flagellar Systems; J Bacteriol. 2007 October; 189(19): 7098–7104. - doi: 10.1128/JB.00643-07
PMCID: PMC204520





Nenhum comentário:
Postar um comentário