A debugagem - Parte 3
7
De utilidade para meus amigos das biológicas:
7.1.
7.1.
7.2.
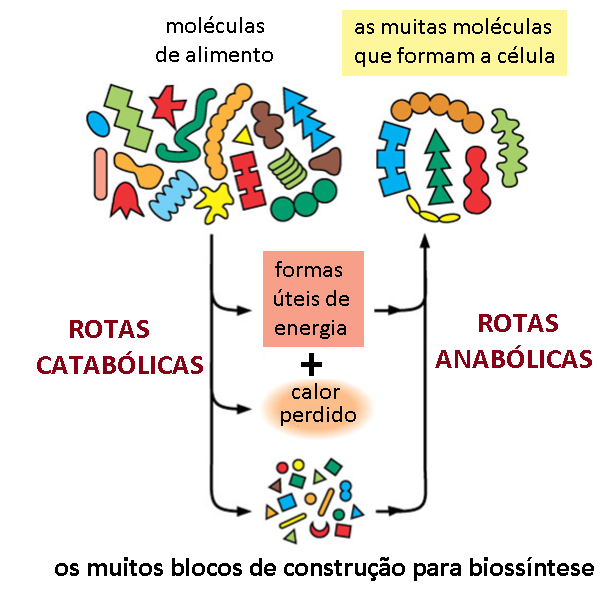
7.3.
Viram (meus amigos das biológicas), como eles tem sua utilidade?
Até material didático nos fornecem!
Até material didático nos fornecem!
8
Outro exemplo do vício que já detectamos:
Toni Gabaldón, Juli Peretó, Francisco Montero, Rosario Gil, Amparo Latorre, and Andrés Moya; Structural analyses of a hypothetical minimal metabolism; Philos Trans R Soc Lond B Biol Sci. 2007 Oct 29; 362(1486): 1751–1762. - www.ncbi.nlm.nih.gov
Abstract
Integramos dados de genômica comparativa e estudos de eliminação de grande escala, num já anteriormente proposto conjunto de genes mínimo compreendendo 206 genes codificadores de proteínas. Para avaliar a consistência do metabolismo codificadas por um tal genoma, mínimo, temos realizado uma série de análises computacionais. Em primeiro lugar, a topologia do metabolismo mínimo foi comparada com a das redes reconstituídas a partir de genomas bacterianos naturais. Em segundo lugar, a robustez da rede metabólica foi avaliada através de mutagênese por simulação e, finalmente, a consistência foi avaliada estequiometricamente derivando automaticamente as soluções de estado estacionário a partir do conjunto de reação. Os resultados indicaram que o metabolismo mínimo proposto apresenta consistência estequiométrica e que é organizado como uma rede de lei de potência complexa com parâmetros topológicos que caem dentro do intervalo esperado para um metabolismo natural de seu tamanho. As análises de solidez revelaram que a maioria das mutações aleatórias não alteram a topologia da rede de forma significativa, mas não causam danos significativos, ao impedir a síntese de vários compostos ou comprometem a consistência estequiométrica do metabolismo. As implicações que estes resultados têm sobre as origens da complexidade metabólica e o projeto teórico de uma célula mínima artificial são discutidos.
Integramos dados de genômica comparativa e estudos de eliminação de grande escala, num já anteriormente proposto conjunto de genes mínimo compreendendo 206 genes codificadores de proteínas. Para avaliar a consistência do metabolismo codificadas por um tal genoma, mínimo, temos realizado uma série de análises computacionais. Em primeiro lugar, a topologia do metabolismo mínimo foi comparada com a das redes reconstituídas a partir de genomas bacterianos naturais. Em segundo lugar, a robustez da rede metabólica foi avaliada através de mutagênese por simulação e, finalmente, a consistência foi avaliada estequiometricamente derivando automaticamente as soluções de estado estacionário a partir do conjunto de reação. Os resultados indicaram que o metabolismo mínimo proposto apresenta consistência estequiométrica e que é organizado como uma rede de lei de potência complexa com parâmetros topológicos que caem dentro do intervalo esperado para um metabolismo natural de seu tamanho. As análises de solidez revelaram que a maioria das mutações aleatórias não alteram a topologia da rede de forma significativa, mas não causam danos significativos, ao impedir a síntese de vários compostos ou comprometem a consistência estequiométrica do metabolismo. As implicações que estes resultados têm sobre as origens da complexidade metabólica e o projeto teórico de uma célula mínima artificial são discutidos.
9
É de se perguntar o que raios esse está fazendo na lista, além de servir de referência para uma citação acima, em campo de bioquímica de um ser vivo atual, mas um certo interesse profissional me levou a incluí-lo:
Chen Liao, Seung-Oh Seo, Venhar Celik, Huaiwei Liu, Wentao Kong, Yi Wang, Hans Blaschek, Yong-Su Jin, and Ting Lua; Integrated, systems metabolic picture of acetone-butanol-ethanol fermentation by Clostridium acetobutylicum; Proc Natl Acad Sci U S A. 2015 Jul 7; 112(27): 8505–8510. - www.ncbi.nlm.nih.gov
Abstract
O metabolismo microbiano envolve processos complexos, em nível de sistemas implementados através da orquestração de reações metabólicas, regulação genética e estímulos ambientais. Um exemplo clássico de tais processos é a fermentação acetona-butanol-etanol (ABE) por Clostridium acetobutylicum, durante o qual as células convertem fontes de carbono para ácidos orgânicos que são depois reassimilados para produzir solventes como uma estratégia para a sobrevivência celular. A natureza da complexidade e sistemas do processo têm sido amplamente subestimados, tornando desafios em compreender e otimizar a produção de solventes.
Clostridium acetobutylicum - John A. Dutton e-Education Institute
Aqui, apresentamos um quadro computacional de nível de sistema para ABE, fermentação que combina reações metabólicas, regulação gênica, e estímulos ambientais. Nós desenvolvemos o quadro pela decomposição de todo o sistema em três módulos, a construção de cada módulo separadamente, e depois montá-los de volta em um sistema integrado. Durante a construção do modelo, uma abordagem “de baixo para cima” foi usada para ligar eventos moleculares no nível de uma única célula para os eventos no nível da população. O modelo integrado foi capaz de reproduzir com sucesso fermentações ABE do C. acetobutylicum de tipo selvagem (ATCC 824), bem como das suas mutações, utilizando dados obtidos a partir de nossas próprias experiências e da literatura.
Além disso, o modelo confere previsões bem-sucedidas das fermentações com várias perturbações de rede através de aspectos metabólicos, genéticos e ambientais. Desde o embasamento para aplicações, o quadro avança nossa compreensão do complexo metabolismo e fisiologia clostridial e também facilita o desenvolvimento de estratégias de engenharia de sistemas para a produção de biocombustíveis avançados.
Anexos
Fruto de minhas “googladas”:
Amanda K. Fisher, Benjamin G. Freedman, David R. Bevan, and Ryan S. Sengera;; A review of metabolic and enzymatic engineering strategies for designing and optimizing performance of microbial cell factories; Comput Struct Biotechnol J. 2014 Aug; 11(18): 91–99. Published online 2014 Sep 3. doi: 10.1016/j.csbj.2014.08.010 - PMCID: PMC4212277
D Segre, D Vitkup, GM Church; Analysis of optimality in natural and perturbed metabolic networks; Proceedings of the National Academy of Sciences 99 (23), 15112-15117. -
doi: 10.1073/pnas.232349399 - www.pnas.org
Carol E. Cleland and Shelley D. Copley; The possibility of alternative microbiallife on Earth; International Journal of Astrobiology 4 (3 & 4) : 165–173 (2005) - doi:10.1017/S147355040500279X - www.researchgate.net
Energy and Life Part I - www.lavc.edu
David Loakes and Philipp Holliger; Darwinian chemistry: towards the synthesis of a simple cell; Mol. BioSyst., 2009,5, 686-694 - DOI: 10.1039/B904024B - pubs.rsc.org
Jia Niu, Ryan Hili & David R. Liu; Enzyme-free translation of DNA into sequence-defined synthetic polymers structurally unrelated to nucleic acids; Nature Chemistry 5, 282–292 (2013) doi:10.1038/nchem.1577 - www.nature.com
Ching-Hsuan Tsai, Jingyang Chen, and Jack W. Szostak ; Enzymatic synthesis of DNA on glycerol nucleic acid templates without stable duplex formation between product and template; PNAS, September 11, 2007, vol. 104 no. 37 - Full: www.pnas.org Detaque: www.pnas.org




Um comentário:
Amei, exatamente o que procurava sobre cirurgia bariatrica, obrigada
Postar um comentário